Журнал «Здоровье ребенка» Том 18, №6, 2023
Вернуться к номеру
Розвиток моноорганних та поліорганних фенотипів бронхіальної астми у дітей: роль комбінованих однонуклеотидних варіацій
Авторы: Дитятковський В.О., Кривуша О.Л., Токарєва Т.М.
Дніпровський державний медичний університет, м. Дніпро, Україна
Рубрики: Педиатрия/Неонатология
Разделы: Клинические исследования
Версия для печати
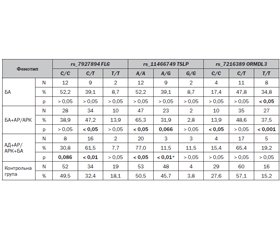
Актуальність. Бронхіальна астма (БА) у дітей посідає провідне місце у структурі захворюваності і смертності серед інших алергологічних і атопічних захворювань. Вона може сформуватися у вигляді моноорганного фенотипу (МОФ), а може і в поліорганному фенотипі (ПОФ) з іншими нозологіями атопічного маршу (АМ) — атопічним дерматитом (АД) та алергічним ринітом/ринокон’юнктивітом (АР/АРК). Цей процес є генетично детермінованим, велику роль у ньому відіграють однонуклеотидні варіації (англ. single nucleotide variations — SNV) генів філагрину (FLG), тимічного стромального лімфопоетину (TSLP) та орсомукоїд-1-подібного білка 3 (ORMDL3). Метою цього дослідження було виявити вплив комбінацій генотипів SNV rs_7927894 FLG, rs_11466749 TSLP та rs_7216389 ORMDL3 в розвитку МОФ та ПОФ атопічної БА у дітей. Матеріали та методи. У дослідженні взяли участь 121 дитина основної та 105 контрольної групи. Критеріями включення до основної групи були вік від 3 до 18 років, клінічно встановлені та лабораторно підтверджені діагнози МОФ БА, ПОФ БА+АР/АРК та АД+АР/АК+БА. Критеріями включення до контрольної групи були: вік від 3 до 18 років, виключені діагнози БА, БА+АР/АРК та АД+АР/АРК+БА. Усім дітям було проведено зскрібок слизової оболонки рота та полімеразну ланцюгову реакцію в реальному часі з отриманим матеріалом на виявлення варіантів генотипних комбінацій rs_7927894 FLG, rs_11466749 TSLP та rs_7216389 ORMDL3. Отримані результати були оброблені за допомогою наступних статистичних інструментів: регресійного логістичного аналізу з визначенням відношення шансів (ВШ) з 95% довірчим інтервалом (95% ДІ), ROC-аналізу з визначенням площі під кривою та специфічності (Sp) і сенситивності (Se), коефіцієнта кореляції Пірсона (r), точного критерію Фішера (ТКФ), коефіцієнта вірогідності Стьюдента. Значенням вірогідності було встановлено p < 0,05, тенденції до вірогідності — p = 0,05–0,1. Результати. Структура вірогідно найчастіших генотипів у когортах основної групи була такою: С/Т rs_7927894 FLG + С/T rs_7216389 ORMDL3 — БА = 8,7 %; С/Т rs_7927894 FLG + Т/T rs_7216389 ORMDL3: БА = 21,7 %, БА+АР/АРК = 18,1 %, АД+АР/АРК+БА = 15,4 %; С/Т rs_7927894 FLG + А/А rs_11466749 TSLP: БА+АР/АРК = 31,9 %, АД+АР/АРК+БА = 42,3 %. Далі наведені показники впливу генотипних комбінацій на ризик розвитку фенотипів БА щодо контрольної групи. МОФ БА: С/Т rs_7927894 FLG + T/T rs_7216389 ORMDL3: r = 0,299, ВШ = 9,44 (95% ДІ 2,07–43,03), AUC = 0,594 (0,507–0,682), Se/Sp = 21,7/97,1 % (p < 0,001). ПОФ БА+АР/АРК: С/Т rs_7927894 FLG + А/А rs_11466749 TSLP: r = 0,136, ВШ = 1,88 (95% ДІ 0,94–3,74), AUC = 0,560 (0,493–0,626), Se/Sp 31,9/80,0 % (р = 0,071); С/Т rs_7927894 FLG + T/T rs_7216389 ORMDL3: r = 0,260, ВШ = 7,49 (95% ДІ 2,05–27,37), AUC = 0,576 (0,528–0,624), Se/Sp = 18,1/97,1 % (р < 0,001). ПОФ АД+АР/АРК+БА: С/Т rs_7927894 FLG + А/А rs_11466749 TSLP: r = 0,207, ВШ = 2,93 (95% ДІ 1,18–7,31), AUC 0,612 (0,507–0,716), Se/Sp = 42,3/80,0 % (р < 0,05); С/Т rs_7927894 FLG + С/T rs_7216389 ORMDL3: r = 0,173, ВШ = 2,50 (95% ДІ 0,99–6,30), AUC = 0,592 (0,489–0,695), Se/Sp = 38,5/80,0 % (р < 0,05); С/Т rs_7927894 FLG + Т/T rs_7216389 ORMDL3: r = 0,222, ВШ = 6,18 (95% ДІ 1,29–29,6), AUC = 0,563 (0,490–0,635), Se/Sp = 15,4/97,1 % (р < 0,01). Відношення асоціацій та ризиків розвитку фенотипів один щодо одного: БА+АР/АРК щодо БА: С/Т rs_7927894 FLG + С/T rs_7216389 ORMDL3: r = 0,171, ВШ = 3,50 (95% ДІ 0,75–16,41), AUC = 0,582 (0,504–0,659), Se/Sp = 25,0/91,3
% (р = 0,095). АД+АР/АРК+БА щодо БА: С/Т rs_7927894 FLG + С/T rs_7216389 ORMDL3: r = 0,345, ВШ = 6,56 (95% ДІ 1,26–34,23), AUC = 0,649 (0,537–0,761), Se/Sp = 38,5/91,3 % (р < 0,05); С/Т rs_7927894 FLG + А/А rs_11466749 TSLP: r = 0,270, ВШ = 3,48 (95% ДІ 0,92–13,17), AUC = 0,625 (0,500–0,750), Se/Sp 42,3/82,6 % (р = 0,059). Висновки. МОФ БА має вірогідну асоціацію з підвищеним ризиком розвитку при генотипній комбінації С/Т rs_7927894 FLG + T/T rs_7216389 ORMDL3. ПОФ БА+АР/АРК має значимі асоціації з підвищеними ризиками розвитку при наступних генотипних комбінаціях: С/Т rs_7927894 FLG + А/А rs_11466749 TSLP та С/Т rs_7927894 FLG + T/T rs_7216389 ORMDL3. ПОФ АД+АР/АРК+БА має найбільше асоціацій з підвищеними ризиками розвитку при наступних генотипних комбінаціях SNV: С/Т rs_7927894 FLG + А/А rs_11466749 TSLP, С/Т rs_7927894 FLG + С/T rs_7216389 ORMDL3, С/Т rs_7927894 FLG + Т/T rs_7216389 ORMDL3.
Background. Bronchial asthma (BA) in children is on one of the leading places in the morbidity and mortality structure among other allergic and atopic diseases. It can be developed in the form of a monoorganic phenotype (MOPh) or a polyorganic phenotype (POPh) with other nosologies of atopic march (AM): atopic dermatitis (AD) and allergic rhinitis/rhinoconjunctivitis (AR/ARC). This process is genetically determined, with single-nucleotide variants (SNV) of filaggrin (FLG), thymic stromal lymphopoietin (TSLP) and orsomucoid-like protein 3 (ORMDL3) genes playing a major role. The purpose of this study was to reveal the impact of rs_7927894 FLG, rs_11466749 TSLP and rs_7216389 ORMDL3 SNV genotype combinations in the development of MOPh and POPh of atopic BA in children. Materials and methods. One hundred and twenty-one children of the main group and 105 controls took part in the study. The criteria for inclusion into the main group were: age from 3 to 18 years, clinically established and laboratory confirmed diagnoses of MOPh BA, POPh BA + AR/ARC and AD + AR/ARC + BA. The criteria for inclusion in the control group were: age from 3 to 18 years, excluded diagnoses of BA, BA + AR/ARC and AD + AR/ARC + BA. All children underwent swabbing of the oral mucosa and real-time polymerase chain reaction with the obtained material to detect variants of rs_7927894 FLG, rs_11466749 TSLP and rs_7216389 ORMDL3 genotype combinations. The results were processed using the following statistical tools: logistic regression analysis with determination of odds ratio (OR) with 95% confidence interval (95% CI), receiver operating characteristic (ROC) analysis with determination of the area under the ROC curve (AUC), sensitivity (Se), specificity (Sp), Pearson’s correlation coefficient (r), Fisher’s exact test, Student’s t-test. The significance value was set at p < 0.05, trend to reliability — at p = 0.0–0.1. Results. The structure of the significantly most frequent genotypes in the cohorts of the main group was as follows: C/T rs_7927894 FLG + C/T rs_7216389 ORMDL3 — BA = 8.7 %; C/T rs_7927894 FLG + T/T rs_7216389 ORMDL3: BA = 21.7 %, BA + AR/ARC = 18.1 %, AD + AR/ARC + BA = 15.4 %; C/T rs_7927894 FLG + A/A rs_11466749 TSLP: BA + AR/ARC = 31.9 %, AD + AR/ARC + BA = 42.3 %. Next, indicators of the genotypic combinations impact on the risk of BA phenotypes development related to the control group are provided. MOPh BA: C/T rs_7927894 FLG + T/T rs_7216389 ORMDL3: r = 0.299, OR = 9.44 (95% CI 2.07–43.03), AUC = 0.594 (0.507–0.682), Se/Sp = 21.7/97.1 % (p < 0.001). POPh BA + AR/ARC: C/T rs_7927894 FLG + A/A rs_11466749 TSLP: r = 0.136, OR = 1.88 (95% CI 0.94–3.74), AUC = 0.560 (0.493–0.626), Se/Sp 31.9/80.0 % (p = 0.071); C/T rs_7927894 FLG + T/T rs_7216389 ORMDL3: r = 0.260, OR = 7.49 (95% CI 2.05–27.37), AUC = 0.576 (0.528–0.624), Se/Sp = 18.1/97.1 % (p < 0.001). POPh AD + AR/ARC + BA: C/T rs_7927894 FLG + A/A rs_11466749 TSLP: r = 0.207, OR = 2.93 (95% CI 1.18–7.31), AUC 0.612 (0.507–0.716), Se/Sp = 42.3/80.0 % (р < 0.05); C/T rs_7927894 FLG + C/T rs_7216389 ORMDL3: r = 0.173, OR = 2.50 (95% CI 0.99–6.30), AUC = 0.592 (0.489–0.695), Se/Sp = 38.5/80.0 % (p < 0.05); C/T rs_7927894 FLG + T/T rs_7216389 ORMDL3: r = 0.222, OR = 6.18 (95% CI 1.29–29.6), AUC = 0.563 (0.490–0.635), Se/Sp = 15.4/97.1 % (p < 0.01). The ratio of associations and risks for developing the phenotypes related to each other: BA + AR/ARC related to BA: C/T rs_7927894 FLG + C/T rs_7216389 ORMDL3: r = 0.171, OR = 3.50 (95% CI 0.75–16.41), AUC = 0.582 (0.504–0.659), Se/Sp = 25.0/91.3 % (p = 0.095); AD + AR/ARC + BA related to BA: C/T rs_7927894 FLG + C/T rs_7216389 ORMDL3: r = 0.345, OR = 6.56 (95% CI 1.26–34.23), AUC = 0.649 (0.537–0.761), Se/Sp = 38.5/91.3 % (р < 0.05); C/T rs_7927894 FLG + A/A rs_11466749 TSLP: r = 0.270, OR = 3.48 (95% CI 0.92–13.17), AUC = 0.625 (0.500–0.750), Se/Sp 42.3/82.6 % (p = 0.059). Conclusions. MOPh BA has a significant association and an increased risk of development with the SNV genotype combination C/T rs_7927894 FLG + T/T rs_7216389 ORMDL3. POPh BA + AR/ARC has significant associations and increased risks of development with the following SNV genotype combinations: C/T rs_7927894 FLG + A/A rs_11466749 TSLP and C/T rs_7927894 FLG + T/T rs_7216389 ORMDL3. POPh AD + AR/ARC + BA has the most associations and increased risks of development within the following SNV genotype combinations: C/T rs_7927894 FLG + A/A rs_11466749 TSLP, C/T rs_7927894 FLG + C/T rs_7216389 ORMDL3, C/T rs_7927894 FLG + T/T rs_7216389 ORMDL3.
бронхіальна астма; фенотипи; діти; однонуклеотидні варіації; філагрин; тимічний стромальний лімфопоетин; орсомукоїд-1-подібний білок 3
bronchial asthma; phenotypes; children; single-nucleotide variants; filaggrin; thymic stromal lymphopoietin; orsomucoid-like protein 3
Для ознакомления с полным содержанием статьи необходимо оформить подписку на журнал.
- Банадига С.В., Волошин С.Б. Генетичні маркери, що визначають виникнення та перебіг бронхіальної астми у дітей. Сучасна педіатрія. 2016. 2(74). 100-104. doi 10.15574/SP.2016.74.100.
- Ntontsi P., Photiades A., Zervas E., Xanthou G., Samitas K. Genetics and Epigenetics in Asthma. Int. J. Mol. Sci. 2021 Feb 27. 22(5). 2412. doi: 10.3390/ijms22052412.
- Ullemar V., Magnusson P.K.E., Lundholm C., Zettergren A., Melén E., Lichtenstein P., Almqvist C. Heritability and confirmation of genetic association studies for childhood asthma in twins. Allergy Eur. J. Allergy Clin. Immunol. 2016. 71. 230-238.
- Mammen J.R., Arcoleo K. Understanding the genetics of asthma and implications for clinical practice. J. Am. Assoc. Nurse Pr. 2019. 31. 384-387. doi: 10.1097/JXX.0000000000000246.
- Волосовець О.П., Досенко В.Є., Кривопустов С.П. [та ін.]. Значення однонуклеотидних поліморфізмів в генах mTOR (rs11121704) та ATG5 (rs510432) в розвитку алергічних захворювань у дітей. Здоровье ребенка. 2015. № 3. С. 5-11.
- Kmyta V., Orlovskyi V., Prystupa L., Prystupa E. Bcl1 polymorphism of glucocorticoids receptor gene and bronchial asthma. Georgian Med. News. 2015 Mar. (240). 51-5. PMID: 25879559.
- Pividori M., Schoettler N., Nicolae D.L., Ober C., Im H.K. Shared and distinct genetic risk factors for childhood-onset and adult-onset asthma: genome-wide and transcriptome-wide studies. Lancet Respir. Med. 2019 Jun. 7(6). 509-522. doi: 10.1016/S2213-2600(19)30055-4.
- Moffatt M.F., Kabesch M., Liang L., Dixon A.L., Strachan D., Heath S., et al. Genetic variants regulating ORMDL3 expression contribute to the risk of childhood asthma. Nature. 2007. 448. 470-473. doi: 10.1038/nature06014.
- Moffatt M.F., Gut I.G., Demenais F., Strachan D.P., Bouzigon E., Heath S., et al. A large-scale, consortium-based genomewide association study of asthma. N. Engl. J. Med. 2010. 363. 1211-21.
- Ferreira M.A.R., Mathur R., Vonk J.M., Szwajda A., Brumpton B., Granell R., et al.; 23andMe Research Team; eQTLGen Consortium; BIOS Consortium; Magnusson P.K.E., Karlsson R., Hinds D.A., Paternoster L., Koppelman G.H., Almqvist C. Genetic Architectures of Childhood- and Adult-Onset Asthma Are Partly Distinct. Am. J. Hum. Genet. 2019 Apr 4. 104(4). 665-684. doi: 10.1016/j.ajhg.2019.02.022.
- Ferreira M.A., Matheson M.C., Tang C.S., Granell R., Ang W., Hui J., et al.; Australian Asthma Genetics Consortium Collaborators. Genome-wide association analysis identifies 11 risk variants associated with the asthma with hay fever phenotype. J. Allergy Clin. Immunol. 2014 Jun. 133(6). 1564-71. doi: 10.1016/j.jaci.2013.10.030.
- Ferreira M.A., Vonk J.M., Baurecht H., Marenholz I., Tian C., Hoffman J.D., et al. Shared genetic origin of asthma, hay fever and eczema elucidates allergic disease biology. Nat. Genet. 2017. 49. 1752-1757.
- Johansson Å., Rask-Andersen M., Karlsson T., Ek W.E. Genome-wide association analysis of 350 000 Caucasians from the UK Biobank identifies novel loci for asthma, hay fever and eczema. Hum. Mol. Genet. 2019 Dec 1. 28(23). 4022-4041. doi: 10.1093/hmg/ddz175.
- Esparza-Gordillo J., Matanovic A., Marenholz I., Bauerfeind A., Rohde K., Nemat K., et al. Maternal filaggrin mutations increase the risk of atopic dermatitis in children: an effect independent of mutation inheritance. PLoS Genet. 2015 Mar 10. 11(3). e1005076. doi: 10.1371/journal.pgen.1005076.
- Дитятковський В.О. Персоналізована генотип-асоційована діагностика прогресування атопічного маршу в дітей. Здоров’я дитини. 2023. Т. 18. № 5. С. 42-50. DOI: https://doi.org/10.22141/2224-0551.18.5.2023.1614.
- Дитятковський В.О., Науменко Н.В., Аліфіренко О.О., Пінаєва Н.Л., Філатова І.А., Таран С.М., Абатуров О.Є. Однонуклеотидні варіанти генів філагрину та глюкокортикоїдних рецепторів у дітей, хворих на різні фенотипи атопічних захворювань. Медичні перспективи. 2022. Т. 27. № 1. С. 132-139. doi: https://doi.org/10.26641/2307-0404.2022.1.254378.
- Marenholz I., Esparza-Gordillo J., Rüschendorf F., Bauerfeind A., Strachan D.P., Spycher B.D., et al. Meta-analysis identifies seven susceptibility loci involved in the atopic march. Nat. Commun. 2015 Nov 6. 6. 8804. doi: 10.1038/ncomms9804.
- Demenais F., Margaritte-Jeannin P., Barnes K.C., Cookson W.O.C., Altmüller J., Ang W., et al. Multiancestry association study identifies new asthma risk loci that colocalize with immune-cell enhancer marks. Nat. Genet. 2018 Jan. 50(1). 42-53. doi: 10.1038/s41588-017-0014-7.
- Andiappan A.K., Sio Y.Y., Lee B., Suri B.K., Matta S.A., Lum J., et al. Functional variants of 17q12-21 are associated with allergic asthma but not allergic rhinitis. J. Allergy Clin. Immunol. 2016 Mar. 137(3). 758-66.e3. doi: 10.1016/j.jaci.2015.08.038.